超分辨率荧光显微镜可用于可视化小于200纳米(即低于光的衍射极限)的结构。显微镜技术之一叫做DNA-PAINT,是由生物化学MPI研究组组长,LMU实验物理教授Ralf Jungmann及其同事开发的。该技术使用短的“成像器”,染料标记的DNA链,它们以互补的方式暂时结合其靶分子,以产生必要的“闪烁”,以超分辨率地重建图像。
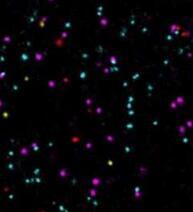
Jungmann说:“最近,我们通过优化DNA序列设计,将DNA-PAINT的传统上较慢的采集速度提高了一个数量级。”“然而,这是以失去多路复用为代价的,这意味着无法同时观察到细胞中的几种结构,” Jungmann补充说。然而,同时观察几种蛋白质对于更好地理解肿瘤与正常细胞之间的复杂信号传导级联非常重要。
在速度优化的DNA-PAINT中无法实现这种多路复用功能,因为只有单一的优化序列具有改进的杂交特性。该论文的第一作者,Jungmann研究小组的同事塞巴斯蒂安·斯特劳斯(Sebastian Strauss)说:“我们问自己如何允许多重成像,同时又进一步提高了图像采集速度。”
在当前的研究中,研究人员提出了一种成功提高成像速度的新颖概念。他们利用了这样一个事实,即成像器与其靶链的结合频率与可用结合位点的数量成线性比例。Strauss说:“结合位点越多,图像获取的速度就越快。但是,简单地串联结合位点将导致不希望的长对接序列,从而可能降低可实现的图像分辨率并增加非特异性结合。”为了规避这些问题,研究人员设计了重复序列基序,例如(TCC)n,可以将其连接起来以提供重叠的结合位点,但链长仅略微增加。“我们设计了六个独立的周期性序列主题,
为了优化新的序列基序并确定其改进基准,该小组使用了DNA折纸结构,这些结构是自组装的纳米尺寸DNA对象,可以自动折叠成预定的形状。这些结构可用于排列DNA-PAINT结合位点,该位点精确间隔开,例如5 nm。这使研究人员能够在确定的条件下评估DNA-PAINT的改进。“新的优化的DNA序列使我们能够在短短几分钟内解析出六个不同的DNA折纸结构,而不仅仅是一个。” Strauss解释说。
“我们很高兴在DNA-PAINT中应用现在进一步提高的成像速度来解决生物学问题。例如,以前只能缓慢地检查肿瘤标志物,而不能在单分子水平上清楚地检查。在我们的研究中,对四个不同的肿瘤标志物证实了它们分子位置和相互作用的快速,准确分析。这可能为药物开发及其作用机理提供重要见解。”
 教育新闻网
教育新闻网